新闻中心
LitVar:关联PubMed 、PMC的基因变异数据语义搜索引擎
![]() 什么是LitVar
什么是LitVar ![]()
大多数生物医学知识在学术出版物中提供非结构化信息。虽然多个数据库提供了有关变异的结构化知识,但它们严重依赖于手动管理,因此需要高级的面向变异的文本挖掘工具来改进注释过程。因此,寻找有关特定基因变异的全面和文本化信息成为一项艰巨的任务,因为研究人员和医疗保健专业人员依赖于不适合多种格式和复杂性的数据库或基于关键字的搜索引擎。所以,迫切需要一个面向变异的语义搜索系统来提高搜索结果的敏感性和特异性。
LitVar是一个可以从生物医学文献中检索变异相关信息,显示变异密切相关基因、疾病和药物信息的搜索引擎。
litvar在变量搜索方面具有多种优势。
首先,litvar使用tmvar,一种高性能的变异名称识别工具,支持摘要和全文文章搜索,它的功能是将同一变异的不同名称规范化为一种独特的标准化形式。这允许返回所有匹配的项目,而不必考虑特定的查询变异名称(例如,对于“A146T”、“C.436G>A”或RS121913527,将返回相同的结果)。
其次,litvar结合了PubMed摘要(>2700万)和PMC全文(>180万)的变异相关文献,并提供了对两种文献资源的统一访问。这一点尤其重要,因为摘要的生物医学概念覆盖率比全文文章低得多。
第三,litvar使用最先进的实体识别工具集作为其后端处理方法,这样用户就可以为感兴趣的变异探索相关的化学和疾病信息。
此外,用户还可以按出版物类型、出版物年份、特定期刊和出版物的不同内容(如摘要或表格内容)进行筛选。除了为用户提供一个用户友好的交互界面外,litvar还支持一组restful api,用于计算分析和对其标准化和规范化的变量数据的开放式编程访问。
Litvar在其数据处理中使用了几个最新的文本挖掘和信息提取工具,如图1所示。
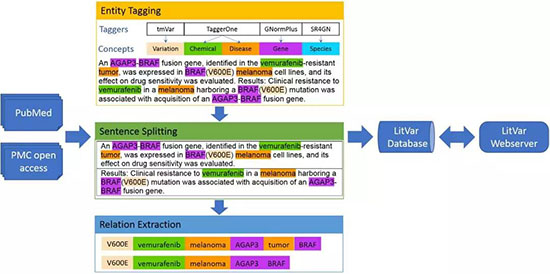
图1. Pre-processing literature data for LitVar. Multiple scripts import publications, detect and normalize biological entities, retrieve relations and continuously update the database.
首先,用Bioc XML格式处理了全部PubMed摘要和PMC全文,然后使用实体标记提取所有变异及其相关实体(即基因、疾病、化学和物种)等信息,实体提取工具包括提取变异的tmvar、提取基因的gnormplus,用于化学品和疾病的TaggerOne和用于物种的SR4GN。然后,我们将所有检测到的实体规范化为相应的数据库标识符。在实体标记之后,应用句子分割器将文章分割成单独的句子。最后,我们根据句子共现性分析实体之间的关系。
![]() 什么是LitVar
什么是LitVar ![]()
工具网址:https://www.ncbi.nlm.nih.gov/CBBresearch/Lu/Demo/LitVar/#
搜索类型包括:核苷酸改变,氨基酸改变(全称和简称),rs号,基因+氨基酸改变(全称和简称)。
图2为搜索结果,多个清晰分隔的区域允许用户执行搜索并可视化结果。这包括a搜索框、b选择和搜索相关其它变异,c过滤器,d实体方面识别的内容,e匹配出版物列表,f位点的基本信息,g颜色区分的基因、基本、变异等,h下载搜索内容。
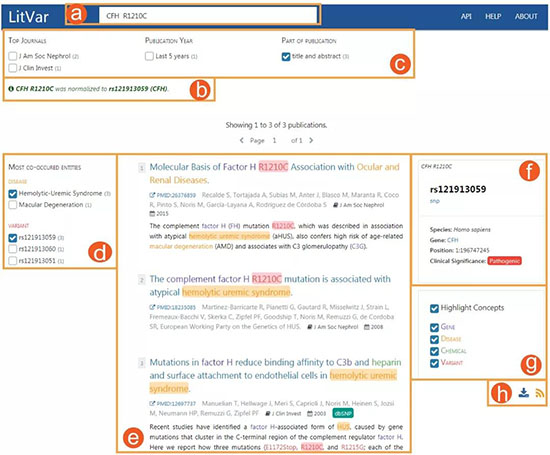
图2:LitVar搜索结果
结论
litvar改进了获取生物医学文献中特定变异信息的途径。litvar不仅处理了全部PubMed摘要,而且还处理了可应用的PMC全文文章。此外,它还允许用户检查其他相关实体,如疾病和化学品。
今后,我们希望通过支持包含其他类型关键实体(如基因和疾病)的查询来扩展litvar的当前范围,并提供基于关键字的查询,同时继续提高litvar在速度和准确性方面的性能。
参考文献:Allot A, Peng Y, Wei C H, et al. LitVar: a semantic search engine for linking genomic variant data in PubMed and PMC[J]. Nucleic acids research, 2018, 46(W1): W530-W536.
