学术资源
跨基因和基因家族的致病性变变异富集区鉴定
在这里,我们比较了基因家族和基因序列中患者错义变异与普通人群错义变异的分布,并开发了一个新的统计框架,根据观察到的变异分布,可以识别蛋白质序列中的致病性变体富集区(PER)。
鉴定的基因家族和基因PER具有高分辨率,可用于变异解释。
本研究中产生的所有数据,包括基因家族比对、PER、变异和旁系保护分数,可访问“PER viewer”
(http://per.broadinstitute.org)。
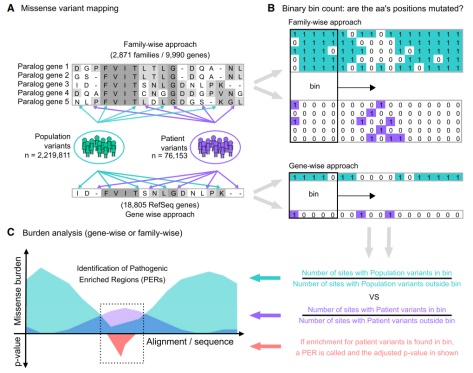
图1.本研究工作流程和PER viewer
(A)从旁系同源基因(基因家族方法)或所有基因(基因方法)的蛋白质比对开始,将gnomAD(普通人群;绿色)和ClinVar/HGMD(患者;紫色)的错义变异独立地定位到相应的氨基酸位置;
(B)映射遵循二进制表示法。对于至少报告了一种错义变异的位点,分配了“1”状态。或者,如果没有发现变异,则注释“0”状态。使用比对/序列上的氨基酸滑动窗口(bin)计数来计算相应的错义变异burden;
(C)窗口内外具有错义变异的位点数量比率定义了burden区域(普通人群burden=绿色;患者burden=紫色);比对序列中普通人群和患者之间变异burden的统计学比较,可以识别出显著的致病性变异富集区(PERs;红色区域)。
基因家族和单基因分析检测到的PER
我们在基因家族和单基因分析中分别鉴定了465个(包含41463个氨基酸)和251个PER(2639个氨基酸)。与基因序列分析方法相比,基因家族方法能够识别更多、更大的PER。
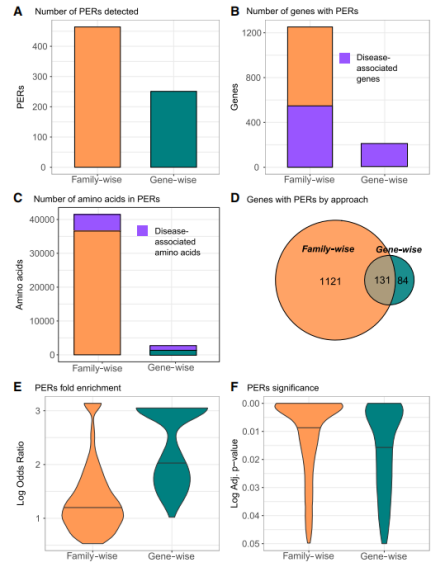
图2.通过基因家族和单基因burden分析检测到的PER
(A)显示了检测到的PER数量;
(B)具有PER的基因数量;
(C)参与PER的氨基酸数量的基因家族(橙色)和基因(绿色)方法的汇总统计数据;
对于B和C,与疾病相关的基因和氨基酸的数量显示为紫色。
(D)为了反映不同方法检测到的具有PERs的基因,显示了维恩图;
(E、F)E和F分别显示了每种方法中检测到的所有PER的总体富集度(对数比值比)和显著性(调整后的P值)分布。
电压门控钠通道基因家族示例
我们显示了由10个旁系基因组成的电压门控钠通道基因家族(家族ID:2614)的错义变异burden*分析结果:SCN1A、SCN2A、SCN3A、SCN4A、SCN5A、SCN7A、SCN8A、SCN9A、SCN10A和SCN11A。由2188个氨基酸组成的10个蛋白质序列的比对,错义变异burden分析确定了16个PER,总体而言,在普通人群变异分布下降的区域,患者变异增加,反之亦然。PER10代表了最长的患者变异富集区,从1466位到1509位有44个连续排列的氨基酸位点。
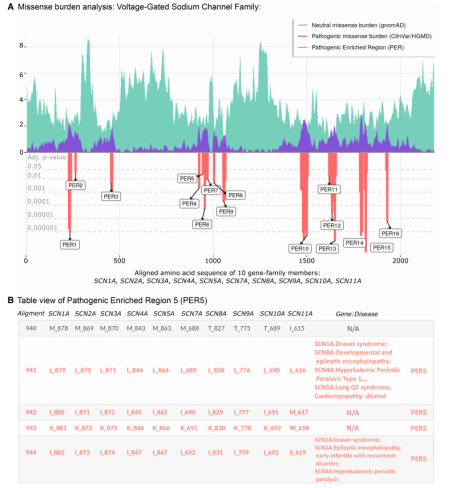
图3. PER viewer示例,电压门控钠通道家族
(A)普通人群和患者错义变异burden分别显示为绿色和紫色。确定的显著致病性变异富集区(PER)显示在红色区域,并与其调整后的P值成比例(灰色水平线);
(B)致病性变异富集区5的列表视图(PER5;位置941-949)。基因列表示在相应氨基酸位置旁边的单个规范序列。“基因:疾病”列显示在患者数据集中观察到的类似疾病。N/A显示排列的氨基酸位置没有疾病报告。
*错义变异burden(Missense burden)是指在一组基因中,某些位置的错义变异的数量或频率。这些位置通常是保守的氨基酸位置,即在不同基因中具有相同或相似的功能和结构。通过对这些位置的变异进行分析,可以研究基因的功能和与疾病相关的变异。
独立数据测试PER
为了在一个独立的数据集中测试PER注释的效用,我们评估了一个大型神经发育(NDD)case-control队列中已确定的PERs内外的新发错义变异(DNVs)的分布情况(Heyne et al.2018)。该数据集包括6753名患者和1911名未受影响的兄弟姐妹,分别鉴定了4404个DNVs和768个DNVs。与对照组DNVs相比,患者DNVs(n=228)在PERs内富集8.33倍(OR=8.33, 95% C.I. =3.90-Inf, P-value=2.72×10−11)。当我们将分析限制在PLI基因(pLI>0.9)时,患者变异在PER中的富集倍数(fold enrichment)甚至更大。
在另一项分析中,我们评估了ClinVar数据库(截止2019年10月)中报告的良性/可能良性和临床意义不确定(VUS)错义变异的分布。我们在PERs中发现了23个良性/可能良性变异和1370个临床意义不确定变异(VUS)。我们注意到,23个良性/可能良性变异中有16个(70%)来自单一提交者,没有一种变异是根据既定的变异解释指南标准进行评估的(Richards et al.2015)。
我们使用来自三个不同时间点的患者错义变异(ClinVar/HGMD)对同一组普通人群变异进行了burden分析:
(1)至2017年12月报告的错义变异(患者变异数= 64,458);
(2)至2018年12月报告的错义变异(患者变异数= 69,863);
(3)至2019年10月(当前;患者变异数=76,153)。
我们观察到相关PER、基因和氨基酸的数量持续增加。
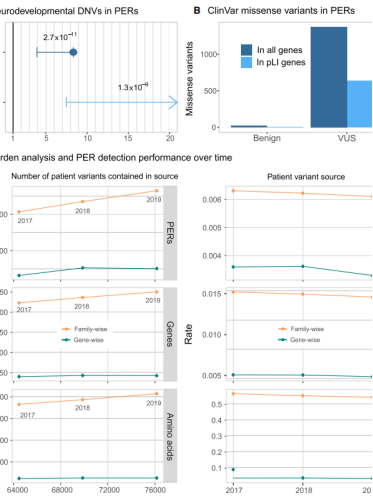
图4.致病性变异在PERs中富集
(A)PERs中的神经发育障碍DNVs。从Heyne et al.(2018)检索到的PERs内DNVs的case-control比较显示了所有基因(蓝色; OR=8.33, 95%C.I.=3.90-Inf, P-value=2.72×10−11)和高概率功能丧失不耐受的基因(浅蓝色; OR=Inf, 95%C.I.=7.48-Inf, P-value=1.34×10−9)。在病例中观察到的Fold enrichment是用one-sided Fisher’s精确检验计算的。在横轴中显示了具有95%置信度的结果比值比(OR)和相应的P值;
(B)PERs内具有良性和未知(VUS)临床意义的ClinVar错义变异(截止2019年10月)的数量。所有基因(蓝色)和仅pLI>0.9基因(浅蓝色);
(C)一段时间内的burden分析性能。对2017年和2018年之前报告的患者变异进行PER检测,并与当前的2019年数据集分析进行比较;
(左)患者变异数量的作为输入函数检测到的PERs、氨基酸和基因的总量;
(右)2017年、2018年和2019年来源中每个患者变异株检测到的PERs、基因和氨基酸的比率。
讨论
当前的工作主要就是通过基因家族分析和单基因方法分析,比对了普通人群和患者人群错义变异分布。在PER检测中,基因家族方法比单基因分析方法更灵敏、更有力。PERs内氨基酸位置的错义变异更有可能被归类为致病性而非良性。这些区域中,患者变异富集,普通人群变异罕见,很可能包括功能上必要的蛋白质特征。研究发现,77.8%的被PERs捕获的氨基酸与保守的功能域重叠,其余的22.2%的位点仍然可以提供额外的生物学见解,暗示了可能不被传统注释直接捕获的新的功能区域。
生成的全外显子PERs地图可以作为变异解释的额外标准。具体而言,PER注释和评估可以被纳入美国医学遗传学和基因组学学院(ACMG)指南的“PM1”类别中。[PM1被定义为“位于突变热点和/或关键且已建立的功能域中的变异,没有良性变异”(Richards等人,2015)。]此外,设计用于检测PERs的统计框架提供了倍增富集和95%置信区间,可以集成到基于ACMG指南的贝叶斯工具中。估计观察到的倍增富集超过18.7可以被视为变异解释的强有力标准。因此,所有PER位点的26.01%可以进一步纳入作为变异解释的强有力标准。
局限性
PER的错义变异burden分析和统计识别在很大程度上取决于用作普通人群和患者数据集参考的变异的数量和质量。我们不能排除PER之外的错义变异是致病的;
属于同一家族的旁系同源可能会发展进化出不同的功能结构域。如果这些结构域富含致病性变体,那么仍然可以识别这些区域的PER。
旁系同源之间的功能冗余并不能保证对变异有相同程度的容忍或不容忍。没有致病性变异的基因会降低在其他家族成员中有致病性变异区域达到显著性的机会。
由于在普通人群中几乎没有限制条件,所以在PER中不太可能发现低外显率或晚发疾病的蛋白质区域。
我们的分析和检测到的PER仅限于本文选取的规范转录本。
在单个蛋白质序列中鉴定功能性必需结构域和位点是罕见孟德尔疾病的挑战,大多数基因注释的患者变异数量仍然很小,限制了变异解释和预测得分的发展,未来较大的变异数据集仍有更多的PER有待确定。
参考文献:
Genome Res. 2020 Jan;30(1):62-71.doi: 10.1101/gr.252601.119. Epub 2019 Dec 23.
