学术资源
临床测序中基因变异分析的最佳实践

Best practices for variant calling in clinical sequencing,于2021年6月10日发表在Developmental Medicine and Child Neurology (IF=5.449) 上。
观点论述
二代测序(NGS)的快速发展加速了遗传病和癌症等领域的研究,并且作为一种先进的基因检测手段应用于临床。随着 NGS 技术的不断发展,也涌现了一些优秀的用于基因变异分析(variants calling)的软件工具。该文章不仅分析了不同测序策略的相对优势和劣势,还针对不同变异类型分享了其在的工具以及分析、验证和基准测试中的实践经验。
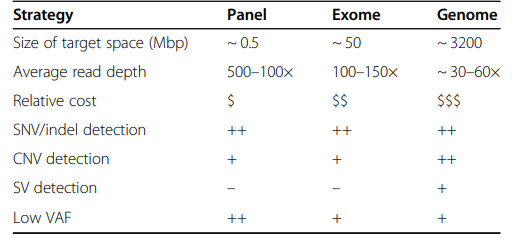
三种测序策略
不同测序策略的选择对variants calling 具有重要的影响,三种测序策略分别为单或多基因的panel、全基因组测序(WGS)和全外显子组测序(WES),每种测序策略都各有利弊。所以测序策略的选择需基于多种因素考虑。
单或多基因的panel
panel测得基因数量少,测序深度最高可达500x,从而可以检测到基因的低频变异,成本低,时间快。
分析具体临床表型相关的基因变异,推荐panel,如OtoSCOPE 听力缺失panel,主要靶向与听力缺失相关的89个基因和microRNA。
Panel的劣势是无法检测基因的结构变异(SV)。
全基因组测序(WGS)
WGS 覆盖整个基因组,平均深度达到30-60x,可以很好的检测出所有类型的变异,并且三种策略中只有WGS才可以检测SV。
但是WGS的成本高,时间久。
全外显子组测序(WES)
WES则介于panel和WGS之间。
所以测序策略的选择是基于多种因素考虑。
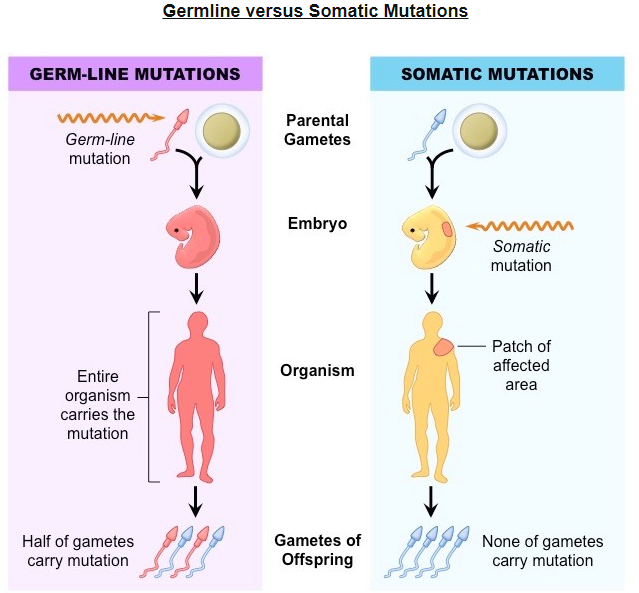
两种变异类型
该文章分别对两种变异类型—胚系突变(Germline Mutation)和体细胞突变(Somatic Mutation)—给出了具体的分析工具和方法。
胚系突变
胚系突变实际是来自上一代的生殖细胞,这种突变会随着胚胎发育一直存在,而在研究方法上,家系分析在胚系突变的分析中占有重要地位。所以在测序过程中,除了先证者个体测序之外,还会加上个体的家属一同测序。
体细胞突变
体细胞突变是不遗传的(非生殖细胞),在研究方法上主要偏重采集癌症组织和正常组织进行比较得到结果。
在检测变异之前,需要做一些预处理。Variant calling 对测序数据的质量要求极高,首先对下机后的数据进行质控,接着将质控后的数据比对到参考基因组,文章中推荐的比对软件是BWA-MEM,比对完之后使用Picard 软件对bam文件进行duplicate 标记,以区分哪些是PCR重复的产物,以便后续分析过滤掉这部分重复数据。GATK 中的模块会对bam文件进行重比对,以纠正mapping时导致的错误(图2A)。
胚系突变分析主要是家系分析,需要指出的一点是,家系的样本在采样和测序的过程中需要完全相同条件,确保检测出的变异不是由于外界干扰导致的。家系分析的样本预处理可以单独进行,而在后续的variant calling时需要联合分析,这样才能判断变异是由遗传导致的还是新生突变。

NGS变异分析的主要步骤及
过程中常用的软件列表
SNV/indel的分析文章中推荐的软件为GATK HaplotypeCaller或Platypus。而CNV和SV分析所利用的软件各有优缺点,需要联合多款软件分析,从而达到灵敏度和特异性的平衡。虽然二代测序技术经过一代测序验证准确率高达99%以上,但因二代测序技术本身还是会产生背景噪音,所以噪音导致的SNVs/indels需要过滤掉。再经过人工使用可视化软件Integrative Genomics Viewer (IGV)对变异进行校验(图3),过滤假阳性。新生突变必须经过sanger测序进行正交验证,而遗传变异不一定需要正交验证,但是至少要满足10个标准,如测序深度, 质量分数, 变异等位基因的序列等(图2B)。
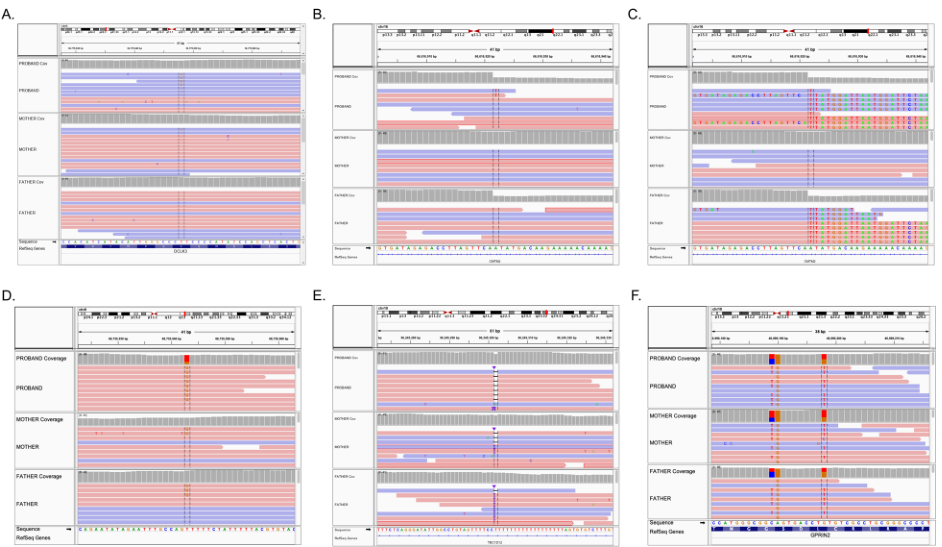
图3 使用IGV可视化软件人工校验变异
评估变异检测的准确性,需要真实变异的数据集来测试软件的准确性。目前最广泛使用的是Genome in a Bottle (GIAB) 和Platinum 数据库中的NA12878 的数据集,这是一个来自欧洲的人类样本,该样本已被世界各地实验室通过各种不同的技术方法进行了测序。
总结
利用NGS数据进行基因变异分析,和NGS技术一样经过十多年的发展,发生了很多变化,仍是一个非常热门的研究领域。NGS 变异分析流程包括比对和预处理、变异分析、过滤噪音及假阳性和人工审查等步骤。有些类型的变异分析只需要单个软件即可,而有些则需要多款软件联合分析。
Koboldt DC. Best practices for variant calling in clinical sequencing. Genome Med. 2020 Oct 26;12(1):91. doi: 10.1186/s13073-020-00791-w. PMID: 33106175; PMCID: PMC7586657.
