学术资源
MitoSAlt:基于NGS数据分析线粒体基因组的缺失和重复



线粒体基因组是独立于核基因组以外的第二套遗传物质,为环状双链DNA分子,全长16 569bp,绝大部分区域是编码区。因此,较大的缺失或重复会严重破坏线粒体基因组的功能;其中线粒体基因组缺失更常见,会导致的癌症、糖尿病、神经性系统疾病等疾病。
线粒体基因组具有多拷贝的性质,单个细胞包括数千个拷贝,因此线粒体变异具有低异质性的特点,这给检测带来了较高的难度。传统检测方法主要是Southern blotting 和 long-range PCR,此法分辨率有限,没法检测所有的变异,且无法准确地区分缺失和重复;而NGS技术可较好地克服这些缺陷。目前已有文献报道了一些检测线粒体大片段缺失和重复的软件,如MitoDel、Splice-Break和eKLIPse等。这些软件一般基于gapped alignments识别缺失,但由于线粒体是环状分子,缺失可能是缺失互补弧的重复,因此这些软件在区分缺失和重复上存在一定局限性。鉴于此,瑞典哥德堡大学Swaraj Basu研究团队开发了基于NGS数据能正确区分线粒体基因组缺失和重复的新软件——MitoSAlt。
MitoSAlt技术原理
MitoSAlt 检测线粒体基因组缺失和重复的原理是基于split alignments。Raw reads与核基因组及线粒体基因组比对,去除比对至核基因组的reads,保留比对至线粒体基因组的reads和unmapped reads,然后重新比对至线粒体基因组。根据split alignments识别缺失和重复并作图。值得注意的是,如图1B所示,线粒体基因组为环形,split read 既可能是缺失,也可能是缺失互补弧的重复。MitoSAlt 对这一情况作了处理,可正确区分缺失和重复。
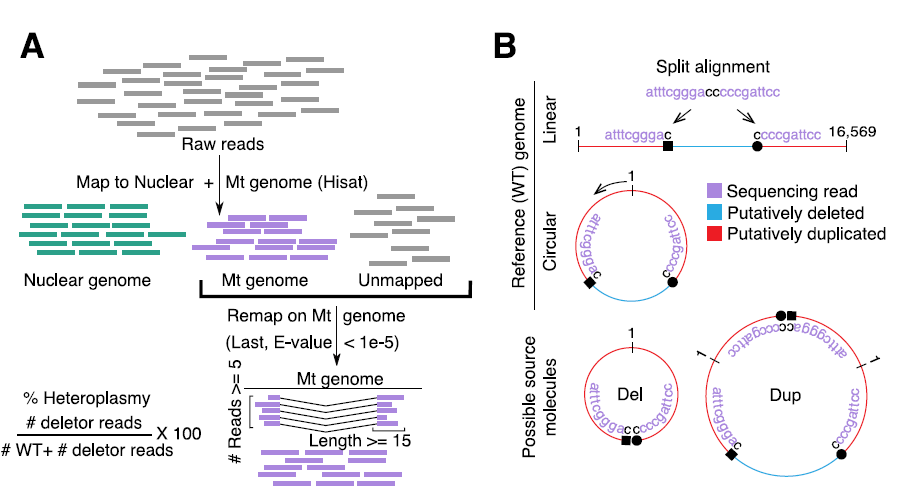
图1 MitoSAlt软件技术原理
MitoSAlt软件性能评估
本文作者共模拟了两次数据,分别是高异质性(16.7%)和低异质性(0.5%)变异。
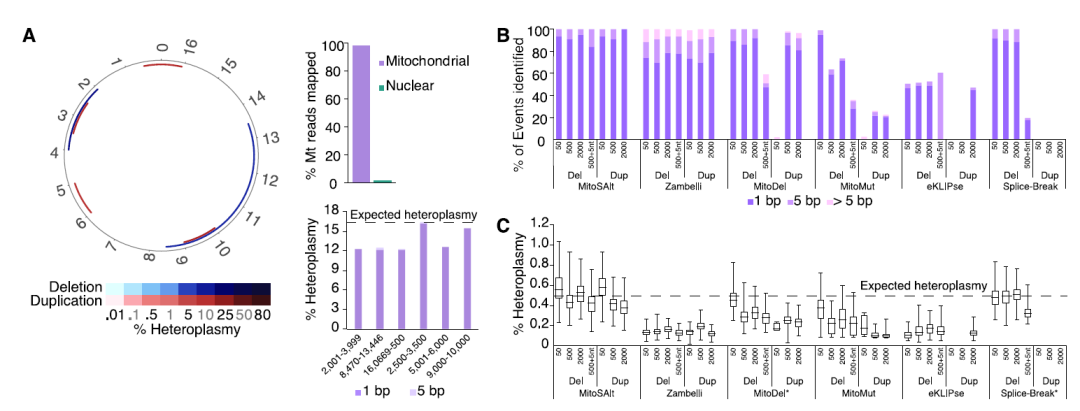
· 高异质性变异数据共有两个缺失(大小分别为2000bp和4976bp)和5个重复(大小都为1000bp),平均覆盖深度约为1600X。MitoSAlt可准确识别到所有变异,并且异质性比率和断点位置都非常精确(图2A)。
· 低异质性变异数据中缺失或重复的大小分布在50、500和2000bp,平均深度约为6000X,MitoSAlt亦可准确到检测到所有变异。
在模拟数据中,MitoSAlt的性能优于Zambelli、MitoDel、MitoMut、eKLIPse、Splice-Break等其它软件(图2B、2C)。
MitoSAlt软件临床真实数据测评
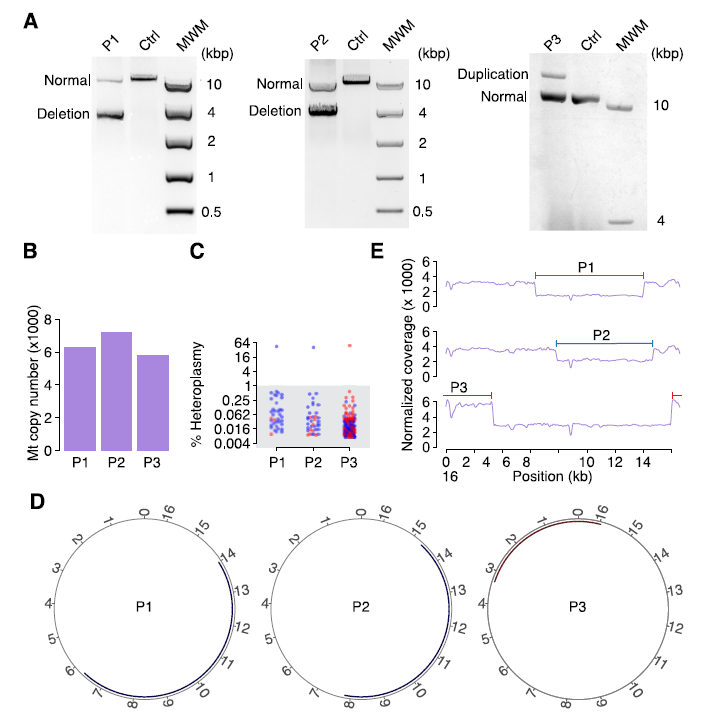
作者用MitoSAlt软件测试了3个真实的病人数据,检测到的变异已经用long-range PCR验证过:2个病人各携带1个缺失,1个病人携带1个重复。3例病人样本都采用WGS测序且平均测序深度在83737X 到 121703X。测试结果显示,MitoSAlt可检测到所有的变异并能正确地区分出缺失和重复;另外,MitoSAlt确定了缺失和重复的断点:缺失区域为 6 330-13 993,和 7 826-14 673、重复在D-loop区域15 973-3 326(图3)。
总结
MitoSAlt是第一款设计区分线粒体缺失和重复的软件。软件主要分析WGS或者富集过MT的样本。经验证,MitoSAlt即可检测到模拟数据中的变异,也可检测到真实病人中的变异,并且正确区分了缺失和重复,且断点判断精确。综合判断,MitoSAlt是一款较为优秀的软件,值得试用。
参考文献:
Accurate mapping of mitochondrial DNA deletions and duplications using deep sequencing[J]. PLoS Genetics, 2020, 16(12): e1009242.
