新闻中心
WisecondorX:低深度全基因组测序(sWGS)检测CNV
研究背景
随着高通量测序价格的降低,低深度全基因组测序(sWGS,0.1-1X)正逐渐成为检测CNV最优秀的常规方法。与芯片(aCGH)相比,sWGS周期短,价格低,测序连续性好,精度高。目前,sWGS已经应用于NIPT、癌症诊断、PGT等项目。
鉴于sWGS的广泛应用,研究者已经开发了不少基于sWGS检测CNV的软件,软件原理都基于读深法(depth of coverage, DOC)。对DOC软件而言,bin值的设定是非常关键的。如果sWGS测序深度较低,bin值应适当增大,bin值越大,噪音越少,但分辨率也越低。DOC软件检测CNV主要包括三步:1.数据标准化, 2.分段(segmentation), 3.异常片段检测。
数据标准化是基础,如果没有标准化,重复序列、GC含量、可映射性(mappability)、多态性等不利因素将对CNV 检测引入非常高的偏差。数据标准化主要有三种方法:1.用健康人的样本建立基线;2.根据参考基因组的已知特征(GC content, mappability等);3.case-control配对样本。本研究根据诊断能力、普及率及有无对照样本等方面选择了六种软件进行比较,如表1所示。
表1 研究选择的软件列表
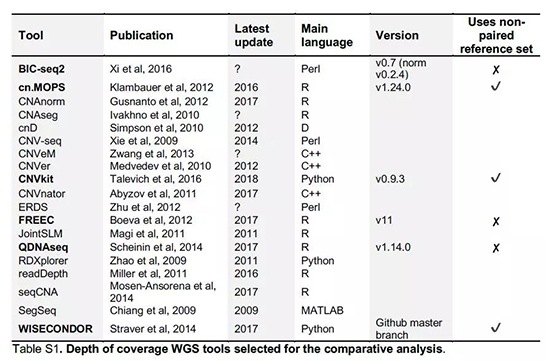
本研究仅比较软件的数据标准化步骤,且采用相同的基因组黑名单(Blacklist),随后用相同的分段方法circular binary segmentation(CBS)检测CNV并比较结果。
材料与方法
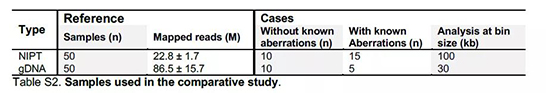
本研究分析了两种测序类型数据NIPT(0.2-0.3X)和sWGS(gDNA,1X),阳性样本共有40个。NIPT样本采用羊穿或绒毛取样验证,分辨率为5Mb;gDNA采用trio分析验证,分辨率约为50-450Kb。
表2 研究使用的样本情况
研究结果
Noise and normality
除了测序深度外,标准化数据理论上可以忽略主要偏差,健康人样本可以保持较好的平整度和正态性。从图1和图2 看出,WISECONDOR处理噪音的能力最强,对健康人样本标准化效果最好,CNVKIT次之。
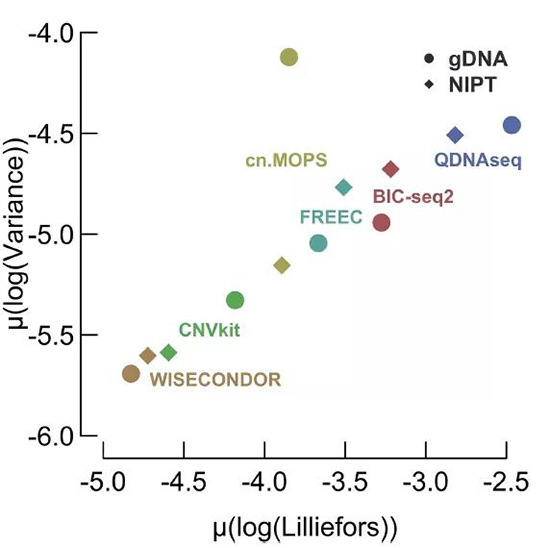
图1 The profile-wide variance versus the Lilliefors normality statistic
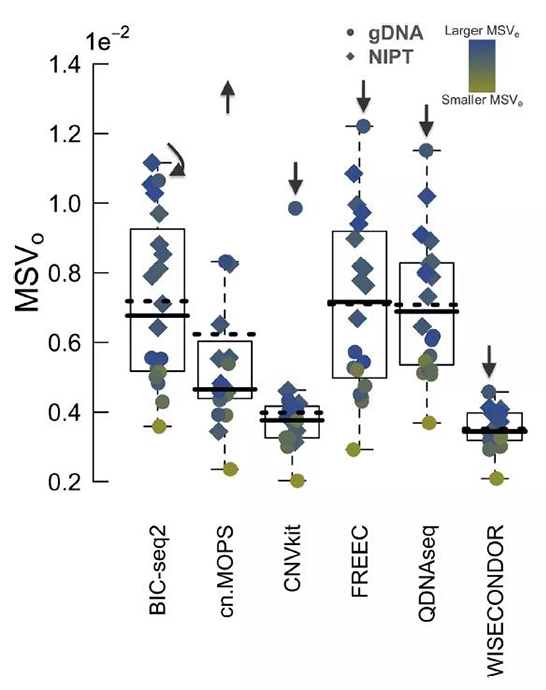
图2 The median segment variance
Performance
六种软件中只有CNVKIT和 WISECON-DOR检测到了所有的阳性CNV,其中WISECONDOR检测到了6个假阳性CNV,CNVKIT检测到了10个。WISECONDOR假阳性率比CNVKIT低。图3 ROC曲线分析显示,WISECONDOR在6个软件中表现最好。
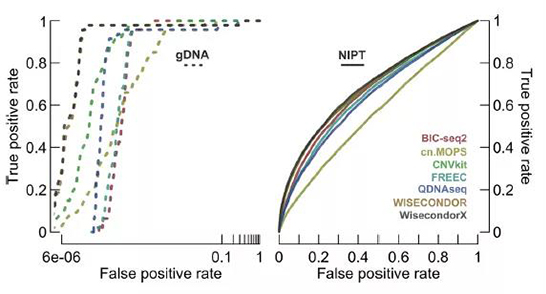
图3 The performance capabilities of normalization techniques
WISECONDOR的局限性及优化
WISECONDOR虽然在数据标准化方面具有较大的优势,但也存在以下局限性:
1、WISECONDOR主要为分析NIPT样本设计,忽视了性染色体;
2、WISECONDOR同时对变异进行分割和评分,bins值小时速度非常慢,平均运行时间24h,分析效率低;
3、WISECONDOR在处理偏差比较大的样本时容易出错。
sWGS样本需分析性染色体,bin值设定小于NIPT样本,且会存在偏差较大的样本。因此,WISECONDOR不太适用于 sWGS样本。针对WISECONDOR的不足,研究人员对软件做了优化,将新软件命名为WisecondorX。
WisecondorX主要做了以下优化:
1、WiseCondorX用高斯混合模型处理性染色体,将男女样本自动分离;
2、Segmentation优化。采用CBS方法,运行时间大幅度降低,分段更准确;
3、Bin值的处理及片段异常处理方法做了改进,检测CNV准确率更高;
4、可视化做了优化。
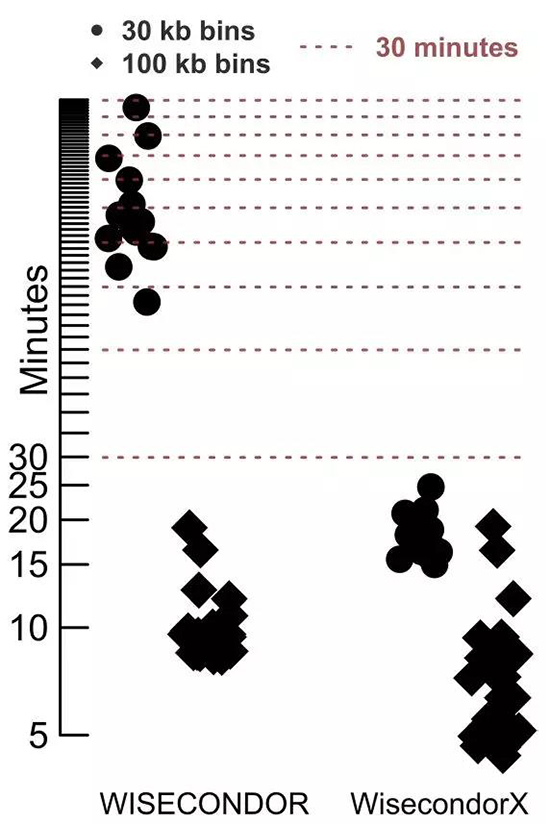
图4 Runtime comparison between WISECONDOR and WisecondorX
讨论
本研究比较了六个软件的标准化方法,在测试中使用了统一的基因组黑名单。WISECONDOR对健康人样本的标准化效果最好,检测到了所有的阳性CNV且假阳性率较低,是表现最佳的软件,CNVKIT次之。CNVKIT和WISECONDOR都实现了样本间的标准化,因此与其它软件相比,样本偏差较小。但WISECONDOR分析sWGS样本具有明显的缺陷,主要表现为无法处理性染色体、耗时长等。WisecondorX针对性解决了这些问题后可用于分析sWGS样本。
参考文献:Raman L, Dheedene A, De Smet M, et al. WisecondorX: improved copy number detection for routine shallow whole-genome sequencing[J]. Nucleic acids research, 2018, 47(4): 1605-1614.
